みなさん、こんばんは!…本当に夜分に失礼します…
ちなみに僕はいつも23時には寝ていまする!
さて!大変お待たせいたしました!!
化学賞予想チーム(田村・高橋・大渕)を代表して、ぶっちーこと大渕が今年のノーベル化学賞を解説します!
今年の受賞者は、Matrin Karplus博士(米・ハーバード大学)、Michael Levitt博士(米・スタンフォード大学)、Arieh Warchel博士(米・南カリフォルニア大学)の3名でした。
彼らの受賞テーマは
「複雑な化学反応系のためのマルチスケールモデルの開発」
です。
彼らによって「古典力学」と「量子力学」を結びつけた計算化学(マルチスケールモデル)の手法が開発され、タンパク質のような大きな分子の構造が変わっていく様子をコンピューターシミュレーションで解明することが可能となりました。その貢献は、創薬から太陽光発電まで非常に多岐にわたります。
さて。
どういうこと???
実は、タンパク質などの生体内の高分子がどのように立体構造を変えながら機能しているのか?ということを調べることは非常に重要なのです。特にタンパク質の場合、正常な立体構造をしていないと、生体内できちんと働かないことさえあります。
ところが、分子の計算を行う場合、どのような分子モデルを扱うかによって計算方法が変わります。
例えば、古典力学に基づいて、ある分子をモデル化するとイラストの左図のようになります。
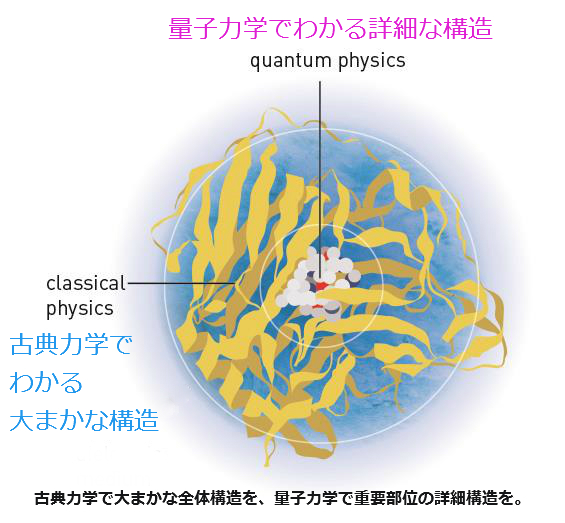
さてさて、 Levitt博士とWarchel博士は、量子力学によるコンピューターシミュレーションを用いた高分子解析手法を開発しました。そして、Karplus博士は、分析機器を用いて実験によってデータを収集し、そこからシュミレーションの元となる計算式を導き出しました。
お三方のこのような功績によって、タンパク質のような巨大な分子も、太陽光パネルの中で光を浴びて電気を生み出す高分子も、コンピューターの中でその動きや形の変化をシュミレーションできるようになったのです。
「高分子の構造決定」という点では、僕の予想 (リンクは削除されました)もいい線行ってたかなと思う(自画自賛)ので残念ですが、受賞されたお三方、本当におめでとうございます(^^)







